Una nueva técnica genómica descubre funciones vitales entre los billones de bacterias que viven en nuestro intestino
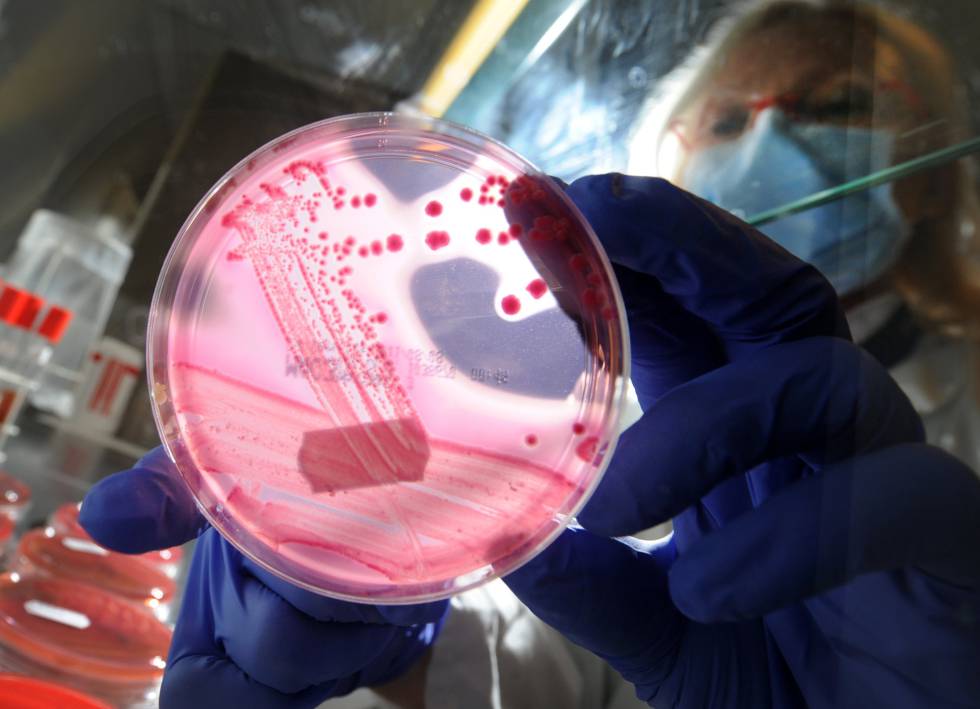
Un cultivo de la bacteria E. coli. CHRISTIAN CHARISIUS AFP/Getty Images
El microbioma (conjunto de microbios que viven en el intestino humano) se ha estudiado hasta ahora aislando y cultivando bacterias por las técnicas convencionales, lo que tiene el problema de que solo algunas bacterias crecen en cultivo. Ahora, el Proyecto Microbioma Humano usa un enfoque metagenómico, consistente en extraer todo el ADN y secuenciarlo en masa, al estilo de lo que se lleva haciendo unos años con el agua de mar. Los científicos empiezan a descubrir así las enzimas y rutas metabólicas que son importantes para los humanos, sobre todo para generar aminoácidos, los componentes de las proteínas.
La investigación del microbioma no solo tiene un interés básico. La composición de nuestras bacterias intestinales afecta a la maduración del sistema inmune humano, y es un factor relevante en el desarrollo de las enfermedades no solo gastrointestinales, sino también cardiovasculares. Sus relaciones con el cáncer y la diabetes están sometidas a investigación activa.

Todo esto quedó atrás y vamos hacia el futuro de la Biología
Emily Balskus y su grupo del departamento de biología química de la Universidad de Harvard, junto a colegas del MIT (Instituto de Tecnología de Massachusetts, también en Boston) y el Instituto Broad, uno de los nodos del proyecto genoma, han desarrollado una nueva técnica llamada enzimología genómica, y presentan sus resultados en Science.
Como todo en biología, las enzimas (proteínas que catalizan reacciones químicas) se agrupan en superfamilias, o grandes grupos de decenas de miles de enzimas relacionadas por su origen común. También suelen compartir aspectos de su mecanismo, pero solo en un sentido profundo de su lógica química que no revela de inmediato su función metabólica concreta, su lugar exacto en el laberinto de reacciones que conforma la cocina de cualquier célula viva. Balskus y sus colaboradores han logrado utilizar los datos masivos del metagenoma de 378 personas, y las poderosas matemáticas de la genómica, para averiguar la función exacta de las enzimas más importantes. Esa es la nueva enzimología genómica.

La técnica funciona de manera similar a la construcción de un árbol genealógico, y produce redes de similitud de secuencia (sequence similarity networks, SSN). Al igual que el ADN, las proteínas son textoso secuencias, que se pueden comparar entre sí como dos frases o dos cadenas de números. Los algoritmos de comparación generan árboles evolutivos, donde cada rama agrupa unas secuencias tan similares que es muy probable que tengan la misma función. Luego hay que comprobarlo por métodos bioquímicos convencionales, pero la enzimología genómica focaliza mucho el problema hasta hacerlo tratable.
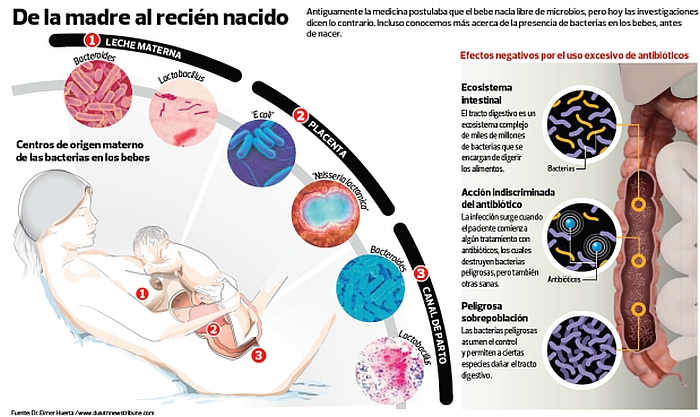
El aparto digestivo está repleto de ellas. Los bebes nacen ya colonizados
Hay otra peculiaridad de las bacterias que resulta de suma utilidad. Los genes de las enzimas con funciones relacionadas, como las que catalizan sucesivas reacciones de la misma ruta metabólica, están a menudo contiguos en el genoma. Conociendo algo de una ruta metabólica, esto da una pista importante sobre la función de cualquiera de ellas.
Para explicar el hallazgo principal de los científicos de Boston, hay que utilizar un poco de nomenclatura bioquímica, que no es el más atractivo de los géneros literarios. Una de las enzimas clave, hasta ahora desconocida, interviene en la ruta metabólica que produce la L-prolina, un aminoácido fundamental en las proteínas: la enzima se alimenta de un compuesto llamado trans-4-hidroxiprolina, lo deshidrata y lo pone en condiciones de generar la L-prolina. Balskus y sus colegas han comprobado que esa enzima es universal en los microbiomas de todas las personas analizadas.
“El metabolismo de la prolina”, escribe en un comentario Margaret Glasner, del departamento de Bioquímica y Biofísica de la Universidad de Texas A&M, “puede ser un nexo importante entre el microbioma del intestino y la salud humana, porque el metabolismo de ese aminoácido se asocia en humanos con el cáncer y con las respuestas celulares al estrés”.
Nuestra química más fundamental
El trabajo abre perspectivas inexploradas hasta ahora. La enzima de la prolina es la más importante caracterizada, pero es miembro de una superfamilia, muchos de cuyos miembros son también ubicuos en todos los metagenomas analizados. Y la técnica, por otra parte, puede utilizarse para descubrir enzimas en otros contextos, como los suelos contaminados y los ambientes extremos.

El microbioma humano se ha revelado en los últimos tiempos como un colaborador necesario de nuestra química más fundamental. Sin él, de hecho, no seríamos seres vivos autónomos. Nuestras bacterias nos ayudan a metabolizar (transformar) los componentes de la dieta que nosotros no sabemos digerir; sintetiza nutrientes y vitaminas esenciales para el funcionamiento de nuestras células (las vitaminas suelen ser coenzimas, o factores que las enzimas necesitan para su funcionamiento); y gestiona compuestos cuyas combinaciones afectan a la salud. La propensión a la obesidad, por ejemplo, tiene relación con la composición del microbioma.
El Proyecto Microbioma Humano es una iniciativa de los Institutos Nacionales de la Salud (NIH) de Estados Unidos, la locomotora de la investigación biomédica pública mundial. Aunque se fundó en 2008 con fondos (115 millones de dólares) para cinco años, sus muestras y resultados siguen siendo una fuente de exploración, como muestra la investigación actual. Su objetivo es el estudio exhaustivo de las bacterias del intestino, sobre todo en relación con las condiciones patológicas. También incluye las bacterias de la boca, la nariz, la piel y la vagina.